我院硕士研究生陈河旭和导师肖传乐研究员及其科研团队在Nature子刊发表最新学术成果
2025年4月4日,肖传乐老师等团队在《Nature Communications》上联合发表了研究论文“Accurate cross-species 5mC detection for Oxford Nanopore sequencing in plants with DeepPlant”(图1),我院研究生陈河旭为该论文的第一作者。这不仅是我院在Nature系列期刊发表成果"零的突破",更是学院AI生态建构、交叉学科培养结出的硕果!

图1. 该文章发表在Nature Communications
DNA甲基化是植物表观遗传调控的核心机制,其在不同序列上下文(CpG、CHG、CHH)的分布特征对基因表达和基因组稳定性具有重要影响,其中CHH基序的甲基化调控在转座子沉默和逆境响应中起关键作用。传统亚硫酸氢盐测序(BS-seq)因短读长限制难以解析重复区域,而纳米孔测序(nanopore sequencing)不仅能提供长读长优势,其原始测序信号能够直接用来检测DNA甲基化修饰,不需要设计额外的生物实验。基于纳米孔测序的植物5mC检测难点主要在于CHH基序,植物中CHH的低甲基化含量(通常仅1-17%)和不同上下文可用于训练模型样本数量的显著性差异,导致极难获得充足的阳性训练数据和高泛化性的CHH甲基化检测模型。随着Oxford Nanopore R10.4芯片的普及,现有甲基化检测工具在跨物种泛化性和CHH位点检测精度上存在显著不足。
为此,肖传乐老师等团队开发了软件DeepPlant,解决了植物CHH型甲基化检测在跨物种泛化性和重复区域解析方面的双重难题,并为作物表观遗传育种和基因组甲基化修饰调控机制解析提供了里程碑式的方法支撑。
该研究首先通过分析十个植物物种的公共BS-seq数据,筛选出CHH高甲基化频率位点(用作训练阳性样本)数量丰富的物种组织(如丹参根组织CHH高甲基化位点占比2.78%),然后通过BS-seq和纳米孔测序构建覆盖97.2% 9-mer上下文的训练集,与之前研究相比显著性提升了(大于三倍)训练样本的上下文覆盖率。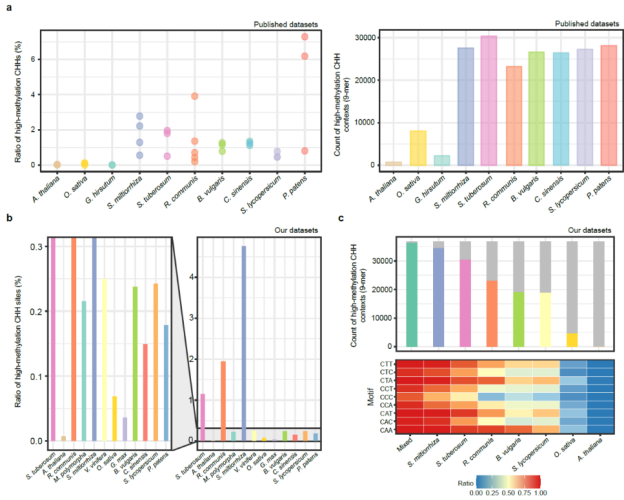
图2. 植物物种高甲基化频率CHH位点数量统计及训练样本筛选
其次,该研究比较了Bi-LSTM与Transformer两种编码器结构以及不同上下文长度对纳米孔测序5mC检测的影响,确认13-mer Bi-LSTM模型在单分子准确率(F1>0.88)与全基因组相关性(r=0.838)间取得最优平衡。DeepPlant还应用了自适应甲基化评分阈值选择方法提高纳米孔甲基化定量与BS-seq的相关性,其应用在Dorado上相比其默认方法对其定量也有显著性提升。综合上述优化,在6个测试物种(包括拟南芥、水稻、番茄、甜橙等)的20倍测序数据集上,DeepPlant展现了不同程度的性能优势(图3);而在研究涉及的全部9种植物中,基于DeepPlant的纳米孔测序全基因组CHH甲基化定量与BS-seq的Pearson相关系数达到0.705-0.838(30倍测序深度),较Dorado提升最高达0.135-0.381。以水稻为例,相关性从0.324跃升至0.705,实现两倍以上提高。相比Dorado和其他纳米孔R10.4甲基化检测软件,DeepPlant在大部分植物物种(7/9)CpG和CHG基序的甲基化检测定量上效果也有提升。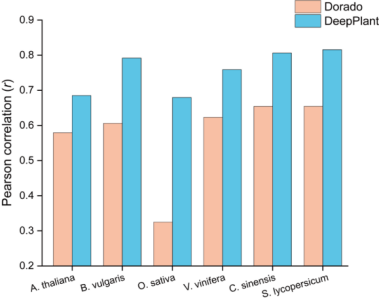
图3. 测试植物物种20×纳米孔测序深度与BSseq定量相关性
最后,利用DeepPlant和纳米孔测序技术,该研究还解析了水稻着丝粒和转座子区域CHH基序的DNA甲基化谱。结果显示,基于约43倍覆盖度的纳米孔长读长数据,DeepPlant对水稻着丝粒区域88%的CHH位点成功进行了甲基化定量,较传统亚硫酸氢盐测序(BS-seq)的37.7%覆盖度提升超两倍,尤其在染色体4、5、8和12的着丝粒区域实现近乎完整的甲基化图谱构建 (图4)。值得注意的是,在着丝粒转座子分析中,DeepPlant首次发现Ac/Ds和Mariner亚类转座子在DNA正链呈现显著甲基化偏好,而LINE和Ty1-copia型转座子则在负链富集,揭示了着丝粒区转座子沉默的机制特征。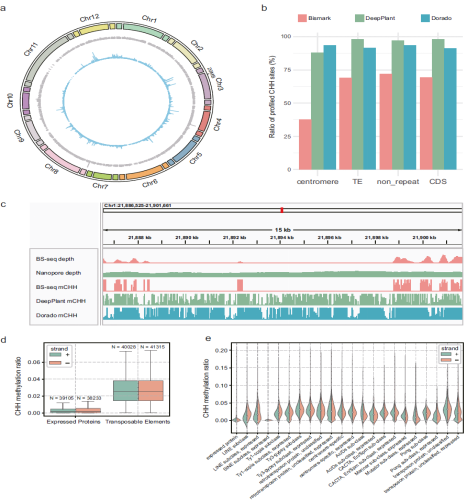
图4. DeepPlant在解析水稻着丝粒区域CHH基序甲基化谱上的应用
文章作者介绍:
第一作者:
陈河旭,中山大学人工智能学院硕士研究生。主要从事高通量测序大数据算法研究,目前以第一作者身份在Nature Communications上发表高水平文章。
通讯作者:
肖传乐,中山大学中山眼科中心研究员(兼职人工智能学院),硕博士生导师,长期从事三代测序前沿技术开发及应用研究。先后开发了三代测序组装方法、修饰识别方法和高级结构识别方法,这些方法成为三代测序数据计算分析重要组成部分,被国内外广泛关注和应用。目前以通讯作者在Nature Methods,Nature Strucutural & Molecular Biology 和 Nature Communications等Nature子刊上发表10余篇论文,获得省部级以上项目10余项,总科研经费达1000余万元。欢迎人工智能学院学生报考研究生,联系方式:xiaochuanle@126.com
中山大学人工智能学院始终以学生成长为中心,培养学生的学习力、思想力、行动力,正在形成跨学科、宽领域、励钻研、促创新的研究生人才培养特色。学院未来将进一步建设涵盖前沿基础突破、学科交叉融合与领域赋能平台等3个层面的丰富AI生态,培养国家所需要的跨学科、复合型、创新型人才。
论文原文:
Chen, HX., Liu, ZD., Bai, X. et al. Accurate cross-species 5mC detection for Oxford Nanopore sequencing in plants with DeepPlant. Nat Commun 16, 5876 (2025). https://www.nature.com/articles/s41467-025-58576-x



